Eine einheitliche und standardisierte Datenauswertung ist die Voraussetzung für die zweckmäßige und effiziente Verwertung von DNA basierten Daten in der Gewässerbeobachtung. Für die Verarbeitung von DNA-Metabarcoding-Daten und die taxonomischen Zuordnung der Sequenzen durch den Abgleich mit Datenbanken (z.B. BOLD und NCBI) gibt es bereits Pipelines und Programme. Bislang fehlt jedoch ein Programm, das eine einheitliche Auswertung der Taxon-Tabellen für Phytobenthos, Makrozoobenthos und Fische ermöglicht, wie beispielsweise die DADA2 Pipeline (https://benjjneb.github.io/dada2/) für Studien mit Mikroorganismen. Das neue Python-basierte Programm „TaxonTableTools“ kann diese Lücke füllen.
TaxonTableTools umfasst fünf individuelle Module zur Prozessierung und Auswertung von Taxatabellen. Die taxonomischen Informationen der DNA-Metabarcoding für TaxonTableTools können einerseits manuell heruntergeladen werden, oder automatisch mit dem Program „BOLDigger“ (Buchner und Leese, 2020), welches einen Abgleich von Sequenzdaten mit der BOLDsystems-Datenbank (http://www.boldsystems.org/index.php) ermöglicht, generiert werden. Diese taxonomischen Informationen können anschließend in das Eingangsformat von TaxonTableTools konvertiert werden. Diese Tabelle beinhaltet Informationen zur Taxonomie der einzelnen Sequenzen als auch die Read-Abundanzen der einzelnen Proben des Projekts. Diese Tabellen dienen als Eingangssformat für alle folgenden Schritte.
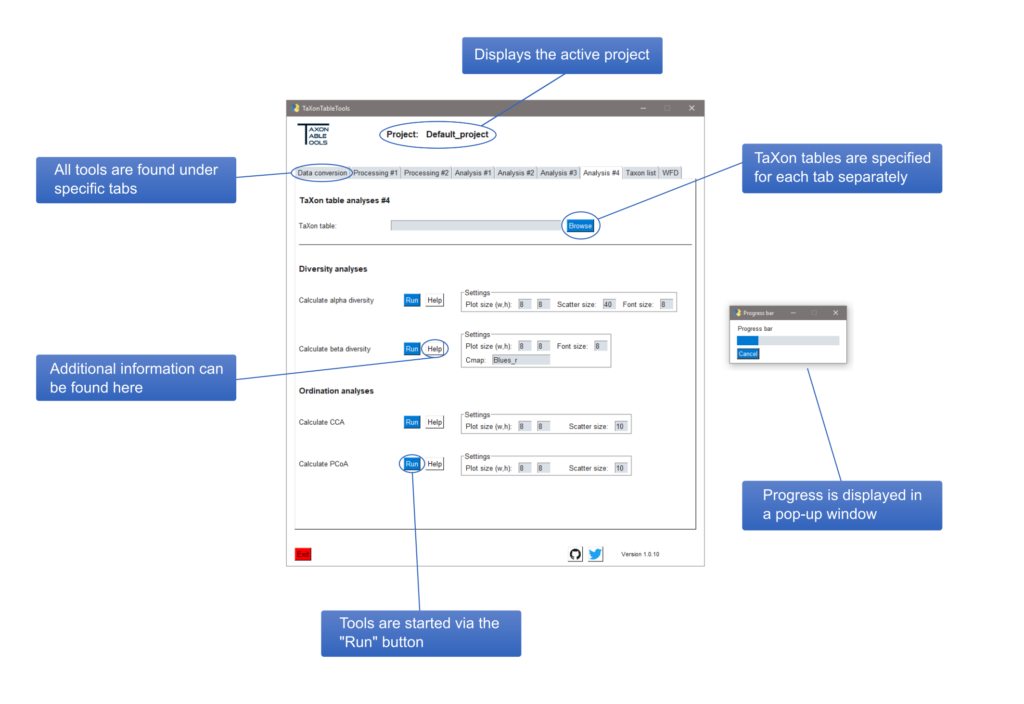
Die Tools der Rubrik „Processing“ erlauben die Zusammenfassung von Replikaten, das Filtern der Proben nach spezifischen Taxa und das Extrahieren ausgewählter Proben aus der Ursprungstabelle. So können die Rohdaten in wenigen Schritten gefiltert und in Untergruppen unterteilt werden.
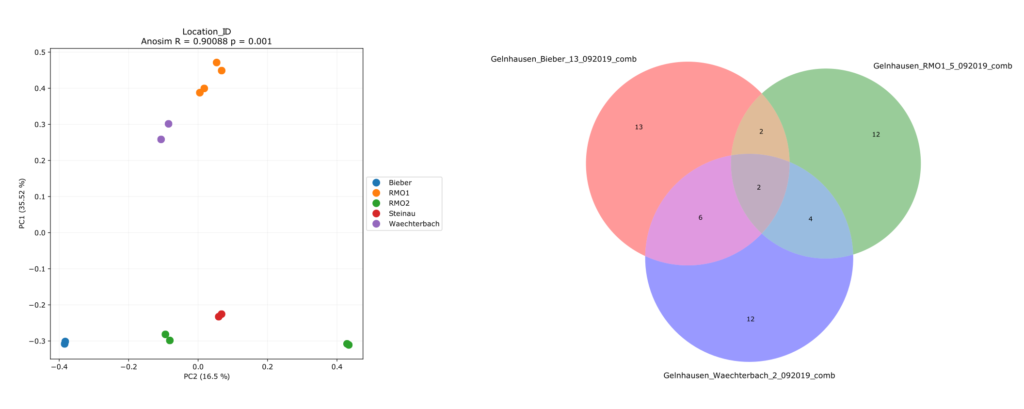
Mit Hilfe der Tools der Rubrik „Analysis“ können verschiedenste Werte und Statistiken auf Basis der Taxon-Tabellen berechnet werden. In der aktuellen Version handelt es sich hier um die taxonomische Auflösung, taxonomische Vielfalt (Abbildung 2.1), OTU-Abundanzen, Venn-Diagramme zum Vergleich von Einzel- und Unterproben, Rarefaction-Kurven (zur Ermittlung des Einfluss der Beprobungsschemas), Read-Abundanzen (Abbildung 2.2), Verteilung der Taxa über die Proben und interaktive Krona-Grafiken (Abbildung 2.3; Bergman und Phillippy, 2011).
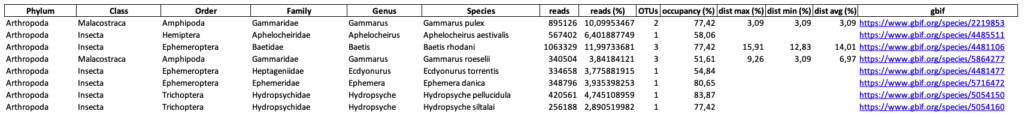
Als Ausgangs-Tabelle können Taxon-Listen erstellt werden, in denen alle Einträge einer Taxontabelle zusammengefasst werden. Auf Wunsch werden die hier aufgeführten Arten gegen die Global Biodiversity Information Facility Datenbank (https://www.gbif.org/) abgeglichen und ein Link zur Datenbank erstellt. Dies erleichtert das Nachschlagen zu biologischen Informationen der Arten enorm.
Zur Bewertung des ökologischen Zustands können die Makrozoobenthos-basierte Taxontabellen im Abgleich mit der offiziellen operationellen Taxaliste (Stand Mai 2011) zu einem mit Perlodes (https://www.gewaesser-bewertung-berechnung.de/index.php/home.html) kompatiblen Eingangs-Format konvertiert werden. In diesem Schritt werden lediglich die An- Abwesenheiten der Taxa berücksichtigt („presence-absence Daten“).
TaxonTableTools ist im Journal Molecular Ecology Resources veröffentlich: TaxonTableTools: A comprehensive, platform-independent graphical user interface software to explore and visualise DNA metabarcoding data (Macher et al., 2021)